9月8日,國(guó)際學(xué)術(shù)期刊Advanced Science發(fā)表了中國(guó)科學(xué)院分子細(xì)胞科學(xué)卓越創(chuàng)新中心(生物化學(xué)與細(xì)胞生物學(xué)研究所)吳薇研究組的研究成果:“Benchmarking Ploidy Estimation Methods for Bulk and Single-Cell Whole Genome Sequencing”。 該研究首次系統(tǒng)評(píng)估了19種倍性檢測(cè)算法在腫瘤基因組分析中的性能,為癌癥臨床診斷和基礎(chǔ)研究提供了工具選擇的關(guān)鍵標(biāo)準(zhǔn)。
染色體數(shù)量變異引起的細(xì)胞倍性異常是癌癥的重要標(biāo)志,與腫瘤演進(jìn)和患者預(yù)后密切相關(guān)。盡管已有多種基于全基因組測(cè)序的倍性檢測(cè)工具,但其性能缺乏統(tǒng)一評(píng)估。該研究通過整合實(shí)驗(yàn)數(shù)據(jù)集和涵蓋不同測(cè)序平臺(tái)、腫瘤純度梯度及測(cè)序深度的模擬數(shù)據(jù)集,對(duì)11種常規(guī)WGS工具和8種單細(xì)胞工具進(jìn)行了全面測(cè)試。研究發(fā)現(xiàn),在腫瘤純度大于30%的常規(guī)樣本中,算法PURPLE展現(xiàn)出最優(yōu)性能,即使在10X低深度測(cè)序條件下仍保持穩(wěn)定;而對(duì)于低純度樣本,PyLOH在純度評(píng)估中表現(xiàn)突出但倍性檢測(cè)能力有限。在單細(xì)胞測(cè)序領(lǐng)域,SeCNV算法在腫瘤細(xì)胞倍性識(shí)別中準(zhǔn)確率最高。值得注意的是,所有現(xiàn)有工具均無(wú)法準(zhǔn)確識(shí)別全基因組加倍所引起的異常整倍體細(xì)胞,且難以適配長(zhǎng)讀長(zhǎng)測(cè)序數(shù)據(jù)。
該研究進(jìn)一步揭示了工具性能的關(guān)鍵影響因素:數(shù)據(jù)預(yù)處理流程可提升TitanCNA等工具的純度評(píng)估精度;基于Strelka的突變檢測(cè)策略在保證準(zhǔn)確性的同時(shí)將效率提升17倍;測(cè)序深度對(duì)Accucopy等工具有顯著制約。這些發(fā)現(xiàn)為腫瘤基因組異質(zhì)性研究、癌癥預(yù)后標(biāo)記物開發(fā)及個(gè)體化治療策略提供了方法學(xué)支撐。
分子細(xì)胞卓越中心博士研究生宋亞偉、梅自律為論文共同第一作者,吳薇研究員與吳樹恒博士為共同通訊作者。研究工作獲得國(guó)家重點(diǎn)研發(fā)計(jì)劃、中國(guó)科學(xué)院戰(zhàn)略先導(dǎo)專項(xiàng)、中國(guó)科學(xué)院國(guó)際伙伴計(jì)劃、國(guó)家自然科學(xué)基金、上海市啟明星項(xiàng)目及上海市科技重大專項(xiàng)的支持,并得到分子細(xì)胞卓越中心高性能計(jì)算平臺(tái)的技術(shù)支撐。
文章鏈接:https://advanced.onlinelibrary.wiley.com/doi/10.1002/advs.202507839
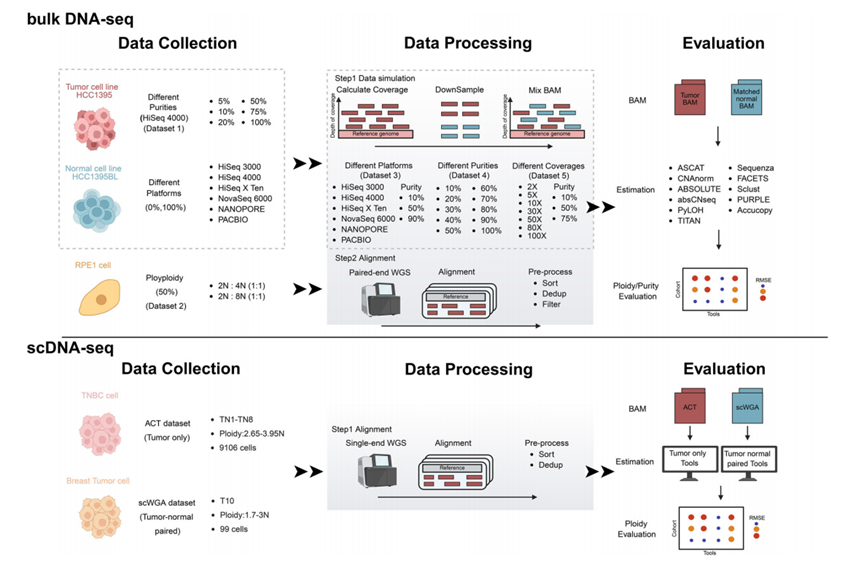
系統(tǒng)評(píng)估工作流程示意圖